-
核小体是真核生物染色质的最小结构单元。每个核小体包括约200 bp的DNA螺旋、一个组蛋白八聚体核心以及一个组蛋白H1分子,其中核心八聚体由H2A-H2B和H3-H4四个异二聚体组成,约147 bp的DNA左手螺旋缠绕于组蛋白核心[1]。核小体之间由DNA连接,形成直径约10 nm的一级串珠结构,再经多次折叠形成更紧密高级结构。紧密的染色质高级结构极大地缩小了核内DNA的空间体积,其动态结构起着调控DNA复制、基因转录和DNA修复等细胞正常活动的重要作用。在电离辐射诱导的DNA损伤修复中,染色质结构发生明显松弛和重塑过程[2-3],以便损伤修复蛋白到达损伤位点。研究表明电离辐射引起的染色质重塑过程与染色质组蛋白乙酰化存在密切联系[4-7]。组蛋白可逆的乙酰化/去乙酰化是一种重要的翻译后修饰,发生在组蛋白N-尾部的赖氨酸残基上,由乙酰转移酶/去乙酰化酶分工完成。修饰的乙酰基会中和组蛋白尾部赖氨酸的正电荷,降低组蛋白与DNA的亲和性,从而使紧密的染色质结构转为疏松结构。Trichostatin A (TSA) 是一种去乙酰化酶非竞争性可逆抑制剂,通过抑制去乙酰化酶活性增加组蛋白乙酰化水平,使染色质结构解聚,同时可延缓细胞周期[8-10]。
染色质结构与分布一般在纳米尺度,传统光学显微镜受衍射极限原理限制,无法达到这一分辨率。随机光学重构显微镜(Stochastic Optical Reconstruction Microscopy, STORM)根据具有光开关功能的荧光分子特性,利用高强度激光使荧光分子快速淬灭与激活获得荧光位置信息,拟合可得到纳米级高分辨率图像[11-12]。Wu等[2]和Zessin等[13]的前期工作表明,利用EdU高密度标记DNA,通过“点击化学”反应将荧光基团掺入DNA,STORM成像技术可以研究染色质精细结构,分辨率优于20 nm。
但是,关于染色质的前期报道多基于染色质结构的形态变化,如何对染色质结构的致密度进行量化分析的研究却较少。本文在STORM技术获得染色质结构的单分子超分辨图像的基础上,结合单分子图像分析算法和重离子辐照活细胞成像技术,研究了电离辐射对染色质结构的重塑及其对DNA损伤修复的影响。
-
人宫颈癌细胞HeLa购于中国科学院典型培养物保藏委员会细胞库。HeLa细胞培养使用DMEM培养基(SH30243.01, Hyclone),培养基中添加10% FBS (F8687, Sigma)、1%青霉素-链霉素(C0222, 碧云天),在体积分数为5% CO2的37 ºC的培养箱中培养。
人纤维肉瘤细胞HT-1080购于中国科学院典型培养物保藏委员会细胞库。HT-1080细胞培养使用MEM培养基(SH30265.01, Hyclone),培养基中添加10% FBS、1%非必需氨基酸(SH30238.01, Hyclone)、1%青霉素-链霉素,在体积分数为5% CO2的37 ºC的培养箱中培养。
用于STORM成像的细胞为复苏后第3-10代细胞,细胞种植于超分辨成像专用35 mm玻璃底培养皿(P35G-1.5-20-C, MatTek)中,密度约105细胞每皿。HT-1080细胞培养24 h后加入10 μmol/L EdU同时加入不同浓度TSA(100, 200, 400, 600 nmol/L),继续培养24 h后固定。HeLa细胞种植后培养24 h,加入10 μmol/L EdU,继续培养24 h后进行X射线辐照,孵育30 min后固定,剂量为1 Gy,剂量率188.5 cGy/min (X-Rad225, PRECISION)。
稳定表达GFP-XRCC1融合蛋白的人纤维肉瘤细胞HT-1080获赠于北京大学杨根博士。GFP-XRCC1-HT-1080细胞培养于微束终端活细胞成像专用细胞盘内,使用DMEM培养基,培养基中添加10% FBS、1%青霉素-链霉素, 在体积分数为5% CO2的37 ºC的培养箱中培养。12 h种植后加入不同浓度TSA(100, 200, 400, 600 nmol/L),孵育12 h后更换含10% FBS的活细胞成像液(A14291DJ, ThermoFisher),并进行重离子辐照及活细胞成像。
-
通过在培养基中加入一种胸腺嘧啶脱氧核糖核苷酸类似物EdU(5-ethynyl-2'-deoxyuridine),细胞在DNA复制时将EdU掺入DNA中实现高密度标记。固定后的细胞按BeyoClick Edu-647试剂盒(C0081S, 碧云天)提供说明书进行细胞通透与“点击化学”反应,使细胞DNA连接上Alexa Fluor 647荧光分子。成像时需使用含β-巯基乙醇(β-ME)等还原性巯基分子的成像液[2]。STORM拍摄时使用高强度647 nm激光使大部分荧光分子进入暗稳态,再使用低强度405 nm激光使少数荧光分子回到基态。基态的荧光分子受激发光被电子倍增电耦合相机(Electron Multiplying Charge Coupled Device, EMCCD)捕获,拍摄帧数15 000帧。得到的图像用Insight3软件(由加利福利亚大学Bo Huang博士提供)进行拟合,拟合完成后由自主研发的算法进行图像漂移矫正。
-
HeLa细胞核内DNA的STORM单分子超分辨成像表明,核内染色质的分布存在几十纳米的团簇结构[2]。为了对染色质结构定量表征,定义任一团簇的最近邻距离NND为该团簇与距其最近的团簇结构的质心间距离,它的分布反映了核内染色质的DNA团簇的密度分布。在开源图像处理软件Image J插件FociPicker3D基础上我们提出了一种团簇的最近邻距离分析算法NND Analyzer(Nearest Neighbour Distance Analyzer)。该算法首先用FociPicker3D分析STORM单分子图像,根据获得的荧光分子局域分布密度特征识别出染色质团簇结构[14],然后测量得到这些团簇结构的面积、单分子密度/数量、质心位置等信息,计算所有团簇的NND距离并对间距250 nm内的NND进行分布统计即得到该细胞核内染色质的团簇定量表征。由于图像中的分子数密度可能影响FociPicker3D对团簇结构的识别和分析,我们进行了不同分子数密度的STORM图像模拟和最近邻距离分析,分析证明,在与实验获取图像相同的分子数密度范围内,根据模拟图像所获得的分子团簇识别与最近邻距离分布对染色质结构分析无影响。为保证实验结果可靠性,本工作中所有细胞核的STORM图像的单分子密度均为4.3分子每百像素,所展示的重构单分子图像(图1)均采用相同对比度设置。
-
活细胞辐照和在线显微荧光观察实验在兰州重离子研究装置(Heavy Ion Research Facility in Lanzhou, HIRFL)加速器的高能微束装置的大气辐照终端进行,实验束流为6.5 MeV/u的Kr离子和25 MeV/u的Ar离子,它们在细胞中的线性能量转移LET分别为约5×103 keV/μm和870 keV/μm。实验时将活细胞培养盘置于微束装置的细胞成像系统显微镜样品台,保持细胞培养皿处于37 ºC恒温状态,调整束流强度使成像视野中每细胞辐照约5个离子每秒[15]。辐照前开始采集图像,每10 s采集一次并储存,采集5帧后开始辐照,采集时长共600 s。采集完成后依次选取每帧中细胞核区域、背景区域和荧光聚点区域并测量其荧光强度,按式(1)计算荧光聚点相对荧光强度,得到荧光强度随时间的变化曲线,再根据式(2)进行拟合,计算得出XRCC1募集到损伤位点的速率常数
$ {k}_{1} $ ,公式如下:$$\begin{split} \\ {I}_{\mathrm{r e l}}=\frac{{I}_{\mathrm{f o c i}}-{I}_{\mathrm{b a c k g r o u n d}}}{{I}_{\mathrm{n u c l e u s}}-{I}_{\mathrm{b a c k g r o u n d}}} \text{,} \end{split} $$ (1) $$ {I}_{\mathrm{r}\mathrm{e}\mathrm{l}}={I}_{0}+{I}_{1}\times (1-{e}^{-{k}_{1}t}) \text{,} $$ (2) 其中:
$ {I}_{\mathrm{r e l}} $ 为相对荧光强度;$ {I}_{\mathrm{f o c i}} $ 为荧光聚点内平均荧光强度;$ {I}_{\mathrm{b a c k g r o u n d}} $ 为背景区域平均荧光强度;$ {I}_{\mathrm{n u c l e u s}} $ 为细胞核平均荧光强度;$ {I}_{0} $ 为辐照前聚点位置处XRCC1相对荧光值;$ {I}_{1} $ 为与损伤DNA结合的XRCC1的荧光强度;$ {k}_{1} $ 为XRCC1募集速率常数。 -
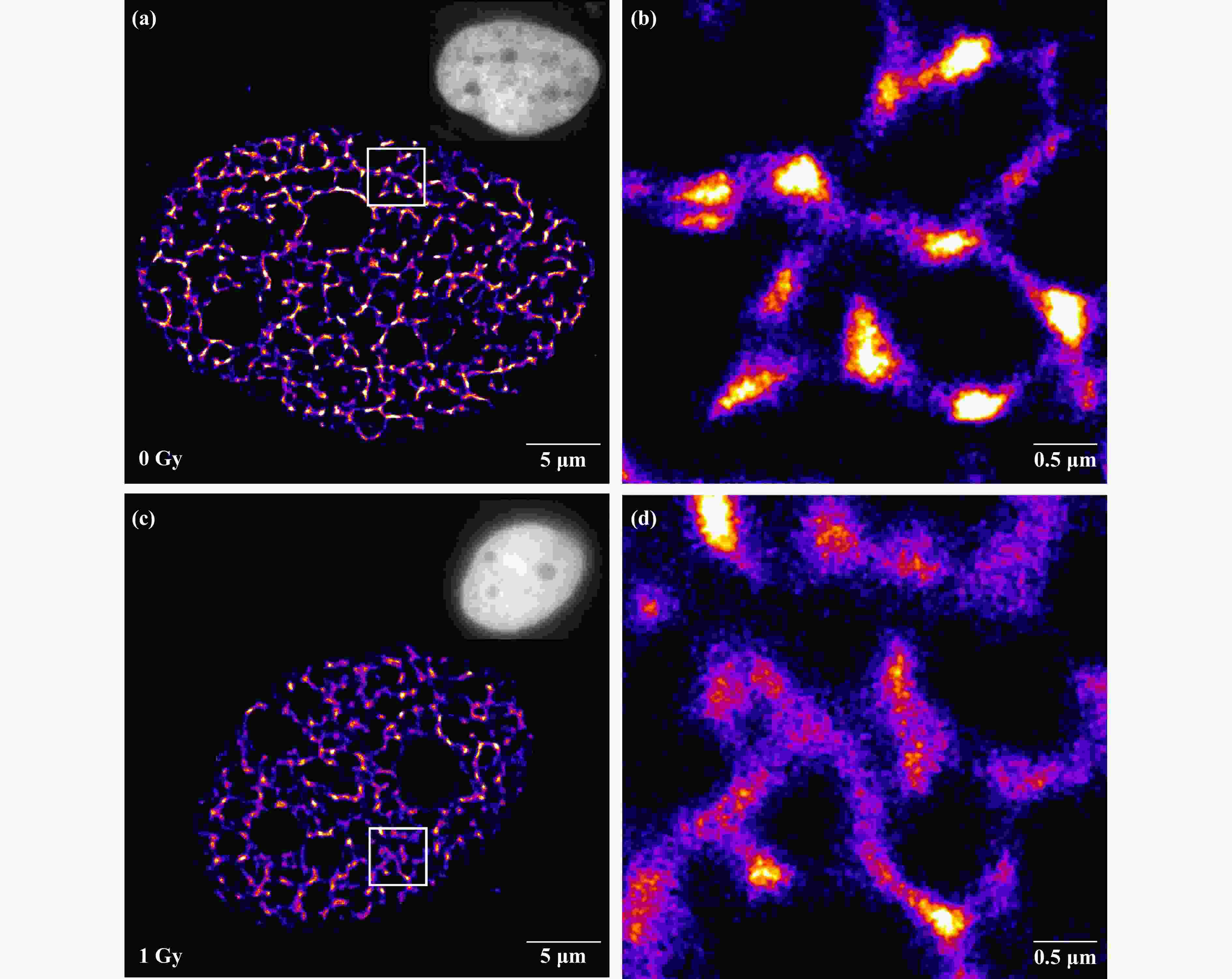
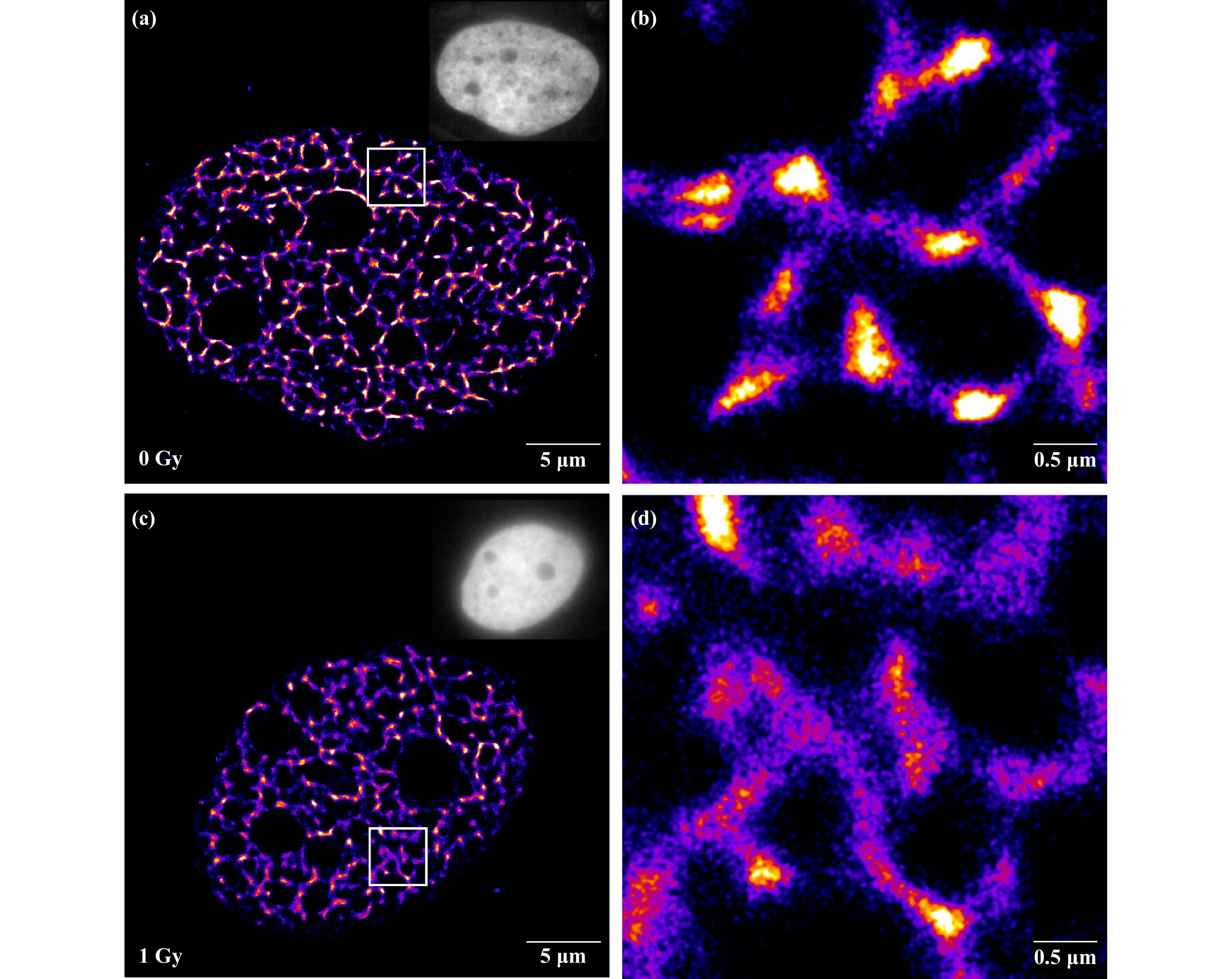
重构后的STORM图像显示了清晰的染色质超分辨结构。区别于宽场显微图像只能看到核区染色质的明亮变化。STORM超分辨图像显示核区由密度不同的DNA区域组成纤维网状结构(图1),纤维结构形状不规则,粗细不一,包围出大的核仁区域及小的空腔区域,内部几乎没有荧光分子。密度高的纤维结构一般比较细,说明染色质结构包装紧密,DNA密度较高。对照组细胞核内纤维粗细200~400 nm,核仁边缘的纤维结构密度相对较高[图1(a)、图1(b)],1 Gy X射线辐照后细胞染色质纤维结构变粗,约为400 nm,核仁周围纤维结构密度降低[图1(c)、图1(d)]。说明1 Gy X射线辐照引起细胞核染色质纤维解开紧密包装,DNA密度变低,染色质结构发生解聚。
-
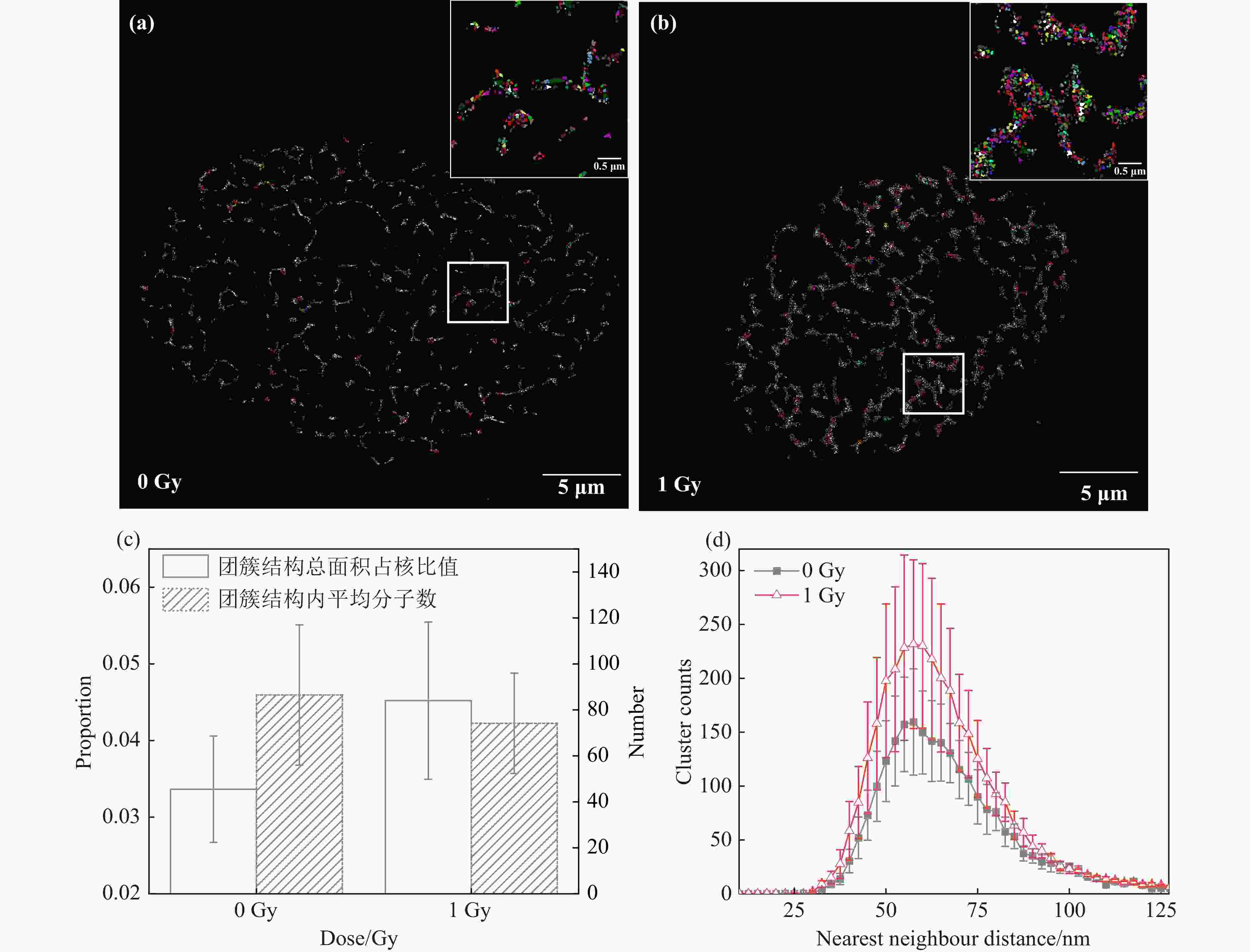
对3.1节中细胞进行纳米团簇结构识别及NND分析,结果显示,0 Gy X射线辐照后细胞染色质团簇结构分布集中,组成粗细不一的不规则纤维网状结构[图2(a)];1 Gy X射线辐照后细胞染色质团簇结构组成的纤维变粗,网状结构间空腔面积变小[图2(b)];团簇结构平均尺寸约55 nm。团簇结构识别结果与图1中染色质结构一致,说明FociPicker3D对染色质团簇结构的识别与划分结果可靠。对这些团簇结构的NND分析结果表明,1 Gy X射线辐照后HeLa细胞中团簇结构总面积相对增加34.3%,团簇结构内包含的平均荧光分子数降低14.3%[图2(c)]。对照组与实验组细胞染色质团簇结构NND分布频数均在55 nm处达到最大值,且1 Gy X射线辐照后细胞内染色质团簇结构数量显著增加[图2(d)]。以上结果表明,X射线辐照引起了明显的核内染色质结构重塑,高密度染色质解聚并向周围扩散,导致可识别团簇结构变多,团簇结构总面积增加,团簇结构内荧光标记DNA密度降低。
-
未经TSA处理的HT-1080细胞染色质团簇结构与HeLa细胞类似,染色质呈现粗细不一的不规则纤维网状结构[图3(a)];经600 nmol/L浓度的TSA处理后HT-1080细胞染色质团簇结构与1 Gy X射线辐照后HeLa细胞染色质团簇结构类似,纤维结构逐渐变粗,纤维密度变低,网状结构空腔面积减小,团簇结构平均尺寸约55 nm [图3(b)]。对0 nmol/L到600 nmol/L TSA浓度处理的HT-1080细胞染色质团簇结构NND分布的分析结果表明,随着TSA浓度增大,染色质团簇结构面积逐渐增加,结构内荧光分子数降低,600 nmol/L TSA处理组细胞比对照组染色质团簇结构数量增加69.1%,团簇结构总面积占核面积比值增加40.1%,团簇结构内平均荧光分子数降低23.0% [图3(c)]。最近邻距离分布曲线在55 nm处达到峰值,且峰值随TSA浓度增加而增大,400 nmol/L后达到最大值[图3(d)]。在100 nmol/L 以下TSA浓度在100 nmol/L处效应与总体趋势不符,这一现象与不同浓度TSA对细胞周期延缓效果有关。以上结果说明TSA处理引起HT-1080细胞染色质结构重塑,高密度染色质结构解聚,该算法可有效表征单分子定位图象中染色质结构特征。
-
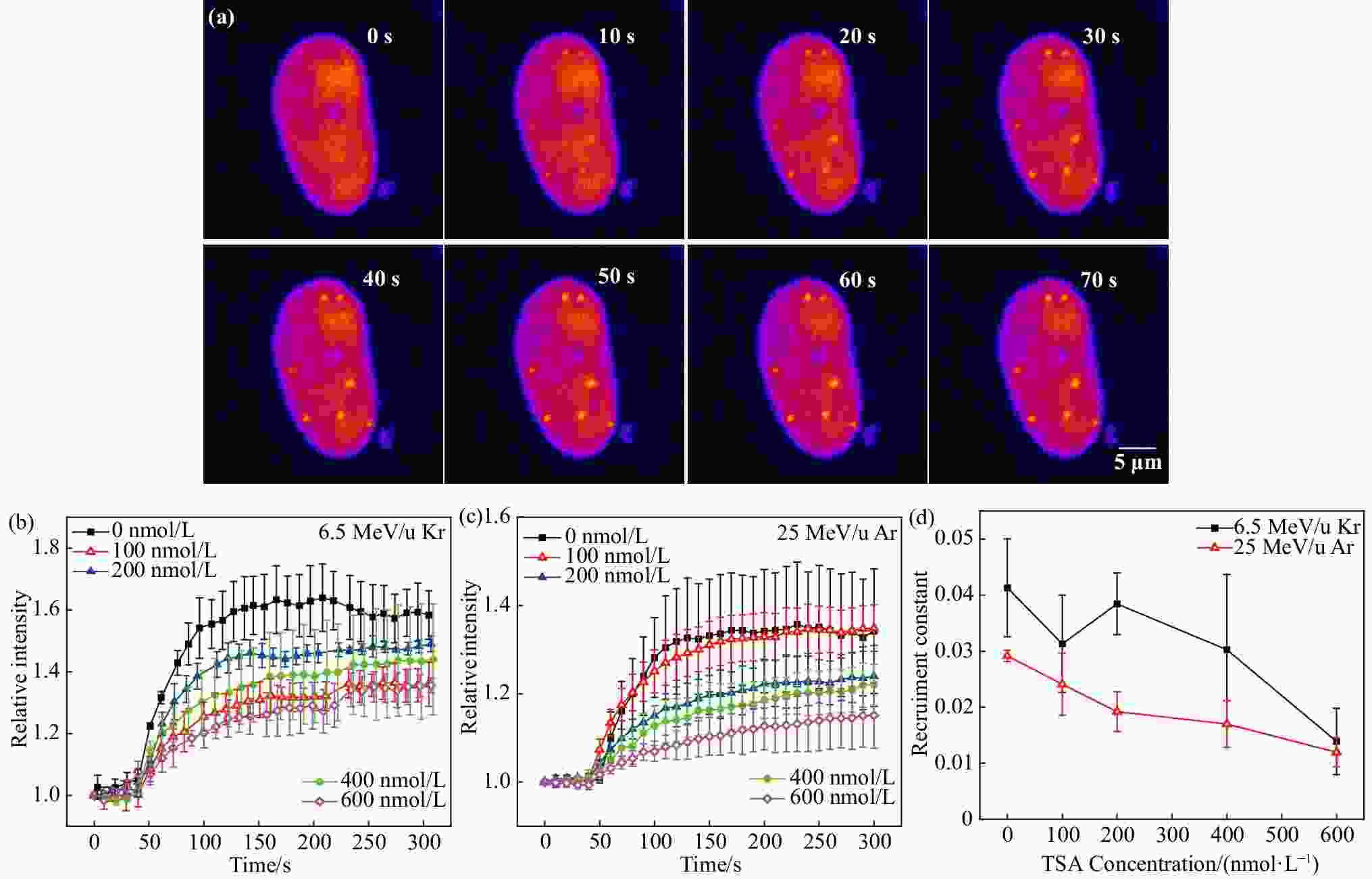
为了研究重塑后的染色质结构对DNA损伤响应和修复的影响,我们利用HIRFL高能微束在线活细胞辐照系统研究了重离子辐照引起DNA损伤处修复蛋白XRCC1(X-ray repair cross complementary protein 1)的蛋白动力学特征。XRCC1是一种重要支架蛋白,在碱基切除修复、单链损伤修复中为多种响应和修复蛋白提供结合位点。经0到600 nmol/L 浓度TSA处理的HT-1080细胞经6.5 MeV/u的Kr离子和25 MeV/u的Ar离子辐照,引起离子径迹内细胞DNA损伤,带有荧光标记的DNA修复蛋白XRCC1被迅速招募到DNA损伤位点处进行修复,活细胞成像系统记录下招募过程。分析表明,在损伤发生后数秒内修复聚点形成,之后亮度持续增加[图4(a)],6.5 MeV/u的Kr离子辐照的细胞在150 s时相对荧光强度达到最大值,25 MeV/u的Ar离子辐照的控制组细胞在150 s时相对荧光强度达到最大值;其他浓度组细胞相对荧光强度在300 s内持续增大,且随着TSA浓度增加,相对荧光强度最大值有降低趋势[图4(a)、4(c)];说明TSA处理降低了沿离子径迹的修复蛋白数量,也即降低了染色质DNA密度,使得重离子造成的DNA损伤密度降低。同时,高LET值离子[图4(b)]引起的损伤处修复蛋白相对荧光强度高于低LET值离子[图4(c)],说明高LET值离子引起的DNA损伤更多。0到600 nmol/L TSA浓度处理组细胞相对荧光曲线拟合分析的结果表明,XRCC1招募速率常数随TSA浓度增加而减小[图4(d)],同时,高LET值离子[图4(d)黑色曲线]引起的损伤处修复蛋白修复动力学常数大于低LET值离子[如图4(d)红色曲线]。说明TSA处理降低了XRCC1与Kr离子造成的DNA损伤之间的亲和性,XRCC1的动力学特征与染色质或DNA损伤密度有关。
-
核内染色质结构的原位高分辨解析和其结构变化表征一直受困于显微成像观测分辨率和图像分析技术的限制。本工作通过采用在细胞培养液中加入胸腺嘧啶核苷类似物EdU,使EdU代替胸腺嘧啶脱氧核糖核苷酸掺入细胞核内复制中的DNA来标记核内染色质。通过点击化学荧光标记EdU和STORM单分子定位显微成像,实验得到了纳米分辨率染色质结构图像,实现了高分辨的染色质纤维结构成像。根据前人报道,组蛋白乙酰化和电离辐射能引起染色质结构解聚[16-17]。利用开发的NND Analyzer最近邻距离算法,分析发现X射线辐照和TSA处理后的细胞核内核小体团簇数量显著增加。在1 Gy X射线辐照后或TSA处理后,染色质团簇结构所占细胞核内的面积比相对于对照组增加,且团簇内平均EdU分子数降低。这些结果表明,核内高密度染色质结构由于乙酰化导致了松散变化并向周围空间扩散,进而使得核小体团簇所占空间增大,团簇内平均EdU分子数降低。100 nmol/L浓度TSA处理对细胞内团簇结构数量与分布及对XRCC1招募动力学特征的影响不同于200到600 nmol/L TSA浓度组,可能原因是不同浓度TSA对细胞周期具有不同的延缓效果[16]。此外,组蛋白的低乙酰化在DNA损伤修复应答中呈现双相调节功能[18-19],低浓度TSA导致的低乙酰化水平在染色质结构调控中的作用值得进一步研究。
与此同时,利用6.5 MeV/u的Kr离子和25 MeV/u的Ar离子辐照乙酰化处理的活细胞,在线显微荧光成像实验获得的XRCC1招募动力学速率常数比对照组低,表明沿离子径迹的DNA损伤密度降低,也即乙酰化处理使得活细胞核内的DNA密度降低,染色质结构发生解聚。XRCC1的反应速率常数降低,这表明XRCC1参与重离子辐射造成的DNA损伤的修复过程不同于理想的生化反应模型,因为在理想模型中反应环境不变的情况下反应速率常数不随底物浓度变化而变化。可能原因是TSA处理改变了损伤位点处染色质所处微环境,乙酰化后组蛋白赖氨酸残基上带负电的乙酰基减弱了带负电DNA与组蛋白结合,还阻碍了DNA与其他DNA修复相关因子的结合;不同浓度TSA对细胞周期的阻碍效果同时也影响着细胞DNA的修复过程。除此之外,Poly(ADP-ribose) polymerase-1 (PARP-1)的多聚核糖基化是XRCC1修复DNA损伤的先决条件,TSA处理也会增加PARP1乙酰化水平,同时阻碍修复因子接近损伤DNA[20-21],对XRCC1的招募速率常数产生一定影响。同时,高LET值离子(如图4(d)黑色曲线)引起的损伤处修复蛋白修复动力学常数大于低LET值离子[如图4(d)红色曲线],说明在复杂的细胞核内环境中,高密度DNA损伤对有限的XRCC1分子的竞争结合使得对其亲和性增加,故而在DNA密度降低时表现出反应常数降低的现象。这一结果与Liu等[22]的实验结果相同,Liu等在实验中观察到,在受25 MeV Kr辐照后G2期细胞相比于G1期细胞有更高的招募速率常数
${k}_{1} $ 和更低的解离常数${k}_{2} $ ,因为G2期细胞DNA含量高,染色质包装紧密,XRCC1扩散受限,高密度的DNA损伤对XRCC1的亲和性增加。 -
染色质结构重塑是一个复杂的、多级的调控网络,涉及多种蛋白质修饰的共同作用。本文利用最近邻距离分析算法NND Analyzer分析细胞染色质中的DNA团簇结构致密程度。基于该算法,研究了电离辐射对HeLa细胞核内染色质结构及TSA对HT-1080细胞和染色质结构的影响,并利用活细胞成像技术获得了重离子引起的HT-1080细胞中DNA损伤中修复蛋白XRCC1的蛋白质动力学特征。结果表明组蛋白高度乙酰化是电离辐射引起染色质高密度结构发生解聚的重要原因,染色质解聚后重离子损伤的XRCC1招募速率常数降低。这些结果表明电离辐射和乙酰化处理均导致了染色质结构的松散化,超分辨成像方法和分析算法及其获得的核小体团簇分布规律为染色质结构的松散提供了直接的定量表征数据支持,对于染色质结构及其对电离辐射生物效应的调控研究具有重要的价值。未来,开展不同品质的电离辐射的辐照对染色质结构重塑的影响将有助于探索重离子治癌新机制和空间辐射的健康风险评估。
致谢 本项工作得到了国家自然科学基金委(11975283、U1632271),科技部国家重点研发计划项目(2021YFA-1601400)和兰州重离子加速器国家实验室的支持,作者感谢北京大学杨根博士为实验提供的稳定表达RFP-XRCC1融合蛋白的人纤维肉瘤细胞,同时感谢HIRFL加速器人员为本工作提供的束流支持。
-
摘要: 真核生物的DNA分子经高度压缩以染色质形式存在于细胞核中,染色质动态结构在DNA复制、基因转录和DNA修复等过程中起着重要的调控作用。核内染色质结构的原位高分辨解析和其结构变化定量表征一直受困于显微成像观测分辨率的限制。通过点击化学荧光标记EdU和STORM单分子定位显微成像,实验得到了细胞核内超分辨率染色质结构图像。基于提出的单分子团簇分析和最近邻距离算法分析发现,X射线辐照和TSA处理后的细胞核内核小体团簇数量显著增多,核小体团簇所占细胞核内的面积比相对于对照组增加,且团簇内平均EdU分子数降低。同时,重离子辐照活细胞在线成像实验获得的XRCC1招募动力学速率常数表明乙酰化处理使得DNA损伤密度降低。这些结果表明电离辐射和乙酰化处理均导致了染色质结构的松散化。STORM超分辨成像方法和分析算法及其获得的核小体团簇分布规律为染色质结构的松散提供了直接的定量表征数据支持。Abstract: Eukaryotic DNA molecule is highly compressed and exist in the form of chromatin in the nucleus. The dynamic structure of chromatin plays an important role in the process of DNA replication, gene transcription and DNA repair. In situ high-resolution analysis of chromatin structure and quantitative characterization of its structural changes have been limited by the optical resolution of microscopic imaging. The super-resolution chromatin structure image in the nucleus was obtained by click-reaction labeled EdU and the STORM single-molecule-localization microscopy. Based on the proposed single molecule cluster analysis and nearest neighbor distance algorithm, we found that the number of nuclear nucleosome clusters increased significantly after X-ray irradiation and TSA treatment, the area ratio of nucleosome clusters in the cell nucleus increased compared with the control group, and the average number of EdU molecules in the cluster decreased. At the same time, the rate constant of XRCC1 recruitment kinetics obtained from the on-line imaging experiment of heavy ion irradiated living cells showed that acetylation reduced the density of DNA damage. These results showed that both ionizing radiation and acetylation led to the decondensation of chromatin structure. The imaging method, analysis algorithm and the distribution of nucleosome clusters provide direct quantitative characterization data support for the decondensation of chromatin structure in cell nucleus.
-
图 4 Kr离子辐射引起的荧光聚点动态及修复蛋白XRCC1招募速率常数变化(在线彩图)
(a) 25 MeV/u的Ar离子辐照后细胞内荧光聚点强度随时间变化,重离子随机辐照细胞核各区域,辐照开始时的时间定义为0 s;(b) 6.5 MeV/u的Kr离子辐照后300 s内不同浓度TSA处理下细胞内荧光聚点相对荧光强度变化,已作归一化处理(50 s时开始辐照);(c) 25 MeV/u的Ar离子辐照后300 s内不同浓度TSA处理下细胞内荧光聚点相对荧光强度变化,已作归一化处理;(d) 6.5 MeV/u的Kr离子辐照后不同浓度TSA处理下XRCC1的招募速率常数${k}_{1} $变化(黑色曲线,0 nmol/L组6细胞,100~200 nmol/L组各5细胞,400 nmol/L组6细胞,600 nmol/L组8细胞);25 MeV/u的Kr离子辐照后不同浓度TSA处理下XRCC1的招募速率常数${k}_{1} $变化。(红色曲线0 nmol/L组4细胞,100 nmol/L组9细胞,200 nmol/L组5细胞,400 nmol/L组4细胞,600 nmol/L组5细胞)。
-
[1] KORNBERG R D. Science, 1974, 184: 868. doi: 10.1126/science.184.4139.868 [2] WU R, LIU W, SUN Y, et al. DNA Repair, 2020, 96: 102974. doi: 10.1016/j.dnarep.2020.102974 [3] GOODARZI A A, NOON A T, DECKBAR D, et al. Molecular Cell, 2008, 31: 167. doi: 10.1016/j.molcel.2008.05.017 [4] JIANG X, XU Y, PRICE B D. Federation of European Biochemical Societies Letters, 2010, 584: 2926. doi: 10.1016/j.febslet.2010.05.017 [5] MAROSCHIK B, GÜRTLER A, KRÄMER A, et al. Radiation Oncology, 2014, 9: 1. doi: 10.1186/1748-717X-9-15 [6] NACKERDIEN Z, MICHIE J, BÖHM L. Radiation Research, 1989, 117: 234. doi: 10.2307/3577324 [7] BIRD A W, YU D Y, PRAY-GRANT M G, et al. Nature, 2002, 419: 411. doi: 10.1038/nature01035 [8] YOSHIDA M, HORINOUCHI S, BEPPU T. Bioessays, 1995, 17: 423. doi: 10.1002/bies.950170510 [9] VIGUSHIN D M, ALI S, PACE P E, et al. Clinical Cancer Research, 2001, 7: 971. [10] YOSHIDA M, BEPPU T. Experimental Cell Research, 1988, 177: 122. doi: 10.1016/0014-4827(88)90030-4 [11] RUST M J, BATES M, ZHUANG X. Nature Methods, 2006, 3: 793. doi: 10.1038/nmeth929 [12] BATES M, HUANG B, ZHUANG X. Current Opinion in Chemical Biology, 2008, 12: 505. doi: 10.1016/j.cbpa.2008.08.008 [13] ZESSIN P J, FINAN K, HEILEMANN M. Journal of Structural Biology, 2012, 177: 344. doi: 10.1016/j.jsb.2011.12.015 [14] DU G, DREXLER G A, FRIEDLAND W, et al. Radiation Research, 2011, 176: 706. doi: 10.1667/rr2592.1 [15] GUO N, DU G, LIU W, et al. Review of Scientific Instruments, 2016, 87: 034301. doi: 10.1063/1.4943257 [16] TÓTH K F, KNOCH T A, WACHSMUTH M, et al. Journal of Cell Science, 2004, 117: 4277. doi: 10.1242/jcs.01293 [17] GUI C Y, NGO L, XU W S, et al. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101: 1241. doi: 10.1073/pnas.0307708100 [18] MILLER K M, TJEERTES J V, COATES J, et al. Nature Structural & Molecular Biology, 2010, 17: 1144. doi: 10.1038/nsmb.1899 [19] HSIAO K Y, MIZZEN C A. Journal of Molecular Cell Biology, 2013, 5: 157. doi: 10.1093/jmcb/mjs066 [20] HASSA P O, HAENNI S S, BUERKI C, et al. J Biol Chem, 2005, 280: 40450. doi: 10.1074/jbc.M507553200 [21] LI S, SHI B, LIU X, et al. Frontiers in Oncology, 2020, 10: 573502. doi: 10.3389/fonc.2020.573502 [22] LIU W, WU R, GUO J, et al. Biophysical Journal, 2022, 121: 1493. doi: 10.1016/j.bpj.2022.03.011 -




 下载:
下载:














 甘公网安备 62010202000723号
甘公网安备 62010202000723号